2022-10-04 来源 : 国家自然科学基金委员会
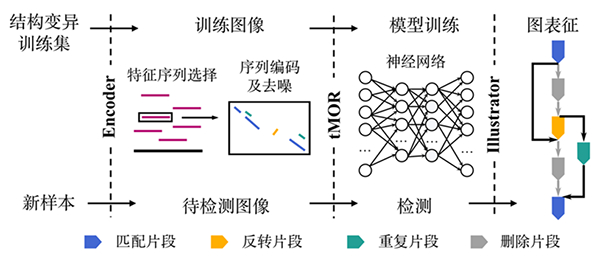 |
图 深度学习实现基于图像的复杂结构变准确检测和表征 在国家自然科学基金项目(批准号:32125009、32070663)等资助下,西安交通大学电信学部自动化学院、第一附属医院叶凯教授团队在基因组复杂结构变异鉴定方法方面取得进展。相关研究成果以“SVision:深度学习方法解析复杂结构变异(SVision: A deep learning approach to resolve complex structural variants)”为题,于2022年9月16日发表在《自然·方法》(Nature Methods)杂志上。论文链接:https://www.nature.com/articles/s41592-022-01609-w。 由于基因组结构变异高发区域序列重复度高、存在大量未知复杂类型,传统基因组学分析基于建模策略的计算方法存在大量错检、漏检,难以深入探究结构变异在生物性状进化和疾病发生中的作用。虽然谷歌公司提出了一种基于深度学习的计算框架,其性能也远超传统检测策略,但面对日益增长的基因组结构变异检测需求,谷歌提出的计算框架只能针对小的插入删除(INDEL)和单核苷酸突变(SNP)进行分析。为开发适用于基因组上大尺度、复杂类型的结构变异的分析方法,叶凯团队提出了一种基于图像序列相似性的编码方法,将基于序列和建模方式的结构变异检测问题转换为基于图像的多目标识别问题,实现了高背景噪声下未知复杂类型结构变异的精准识别与表征(图)。通过使用该方法,研究者系统地分析了单个人类基因组中携带的复杂结构变异,发现复杂结构变异在基因功能和基因组进化中起到重要作用。本项工作为后续利用大规模人群队列研究全类型结构变异奠定了基础。 本项工作通过深度学习策略从头发现基因组复杂结构变异,为基于前沿信息技术的生物大数据计算理论提供了新思路。本项目成果得益于自动化、数学以及生物学的交叉团队协作努力,也为后续交叉合作研究提供了经验。
版权声明:本网站所有注明来源“医微客”的文字、图片和音视频资料,版权均属于医微客所有,非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源:”医微客”。本网所有转载文章系出于传递更多信息之目的,且明确注明来源和作者,转载仅作观点分享,版权归原作者所有。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。 本站拥有对此声明的最终解释权。





发表评论
注册或登后即可发表评论
登录注册
全部评论(0)